opencv-python เบื้องต้น บทที่ ๙: การใช้ตัวกรองเพื่อค้นหาหรือเน้นส่วนขอบ
เขียนเมื่อ 2020/06/28 18:57
แก้ไขล่าสุด 2024/02/22 10:25
ต่อจาก บทที่ ๘
ในบทที่แล้วได้พูดถึงการใช้ตัวกรองเพื่อทำคอนโวลูชันไปแล้ว
บทนี้ก็ยังคงเป็นเรื่องของตัวกรองเช่นกัน แต่จะมาพูดถึงการใช้ตัวกรองเพื่อทำการเน้นขอบ ซึ่งมีอยู่หลายวิธีด้วยกัน
ตัวกรองเพื่อเน้นขอบ
ประโยชน์อย่างหนึ่งของการใช้ตัวกรองทำคอนโวลูชันก็คือการค้นหาส่วนที่เป็นขอบ คือบริเวณที่มีการเปลี่ยนความเข้มสีอย่างชัดเจน
ตัวกรองสำหรับเน้นขอบก็คือตัวกริงที่เมื่อนำมาใช้คอนโวลูชันกับภาพแล้วจะทำให้ได้ค่าตรงบริเวณที่เป็นขอบของภาพเป็นค่าสูงขึ้นมา
มีหลายแนวคิดในการทำตัวกรองในลักษณะแบบนั้น ที่ง่ายที่สุดก็คือตัวกรองเพรวิต (Prewitt filter) ซึ่งมีลักษณะแบบนี้
ในที่นี้ตัวกรอง Kx จะตรวจจับบริเวณที่มีการเปลี่ยนแปลงในแนวนอน (เส้นขอบแนวตั้ง) ส่วน Ky จะตรวจจับบริเวณที่มีการเปลี่ยนแปลงแนวตั้ง (เส้นขอบแนวนอน)
ที่ทำแบบนั้นได้ก็เพราะค่าของตัวกรองเรียงกันตามแนวตั้งหรือแนวนอนอย่างใดอย่างหนึ่ง โดยค่ารวมกันทั้งหมดเป็น 0 ดังนั้นถ้าเจอบริเวณที่ค่าเปลี่ยนอย่างรวดเร็วก็จะเกิดค่าเป็นบวกหรือลบอย่างชัดเจน แต่ถ้าบริเวณไหนไม่มีการเปลี่ยนแปลงก็จะเป็น 0
ลองทดสอบโดยการสร้างอาเรย์ง่ายๆขึ้นมาแล้วสร้างตัวกรองทั้ง ๒ แบบมาใช้ทำคอนโวลูชันด้วยฟังก์ชัน cv2.filter2D() แล้วดูผลที่ได้ด้วย matplotlib
import cv2
import numpy as np
import matplotlib.pyplot as plt
# ตัวกรองแนวนอน
prewittx = np.array([[-1,0,1],
[-1,0,1],
[-1,0,1]])
# ตัวกรองแนวตั้ง
prewitty = np.array([[-1,-1,-1],
[0,0,0],
[1,1,1]])
# ภาพที่ต้องการพิจารณา
arr = np.array([[0,1,1,1,0,0],
[0,1,1,1,1,0],
[0,1,1,1,1,1],
[0,1,1,1,1,1],
[0,1,1,1,1,0],
[0,1,1,1,1,0]],
dtype=np.float32)
plt.figure(figsize=[4,10])
plt.subplot(311) # วาดภาพเดิม
plt.imshow(arr,cmap='gray')
plt.subplot(312) # ใช้ตัวกรองแนวนอน
plt.imshow(cv2.filter2D(arr,-1,prewittx),cmap='jet')
plt.colorbar(pad=0.01,aspect=40)
plt.subplot(313) # ใช้ตัวกรองแนวตั้ง
plt.imshow(cv2.filter2D(arr,-1,prewitty),cmap='jet')
plt.colorbar(pad=0.01,aspect=40)
plt.show()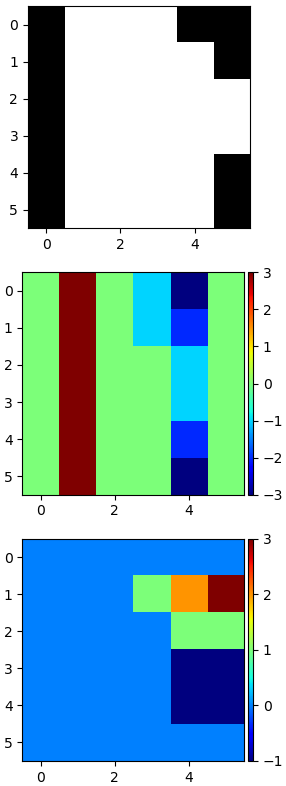
อาเรย์ตัวอย่างในที่นี้มีการเปลี่ยนแปลงอย่างเด่นชัดในแนวนอน ผลลัพธ์จะเห็นว่าตัวกรองแนวนอนให้ค่าเป็นบวกหรือลบอย่างมากตรงบริเวณที่เป็นขอบ
ข้อควรระวังอย่างหนึ่งก็คือ เมื่อใช้ตัวกรองแบบนี้แล้วจะทำให้เกิดค่าติดลบได้ ดังนั้นถ้าข้อมูลภาพเป็น uint8 อยู่ควรเปลี่ยนชนิดข้อมูลเป็น float32 เพื่อให้มีค่าติดลบได้
ส่วนการแสดงผลใน matplotlib เมื่อมีสีเดียวแบบนี้จะแสดงสีโดยไล่จากค่าต่ำสุดไปสูงสุดอยู่แล้ว แม้จะติดลบก็สามารถแสดงผลได้ ในที่นี้ใช้สีรุ้ง พร้อมวาดแถบสีบอกค่าไว้ทางขวาด้วยเพื่อให้เห็นชัด
ลองเปลี่ยนเป็นอาเรย์แบบนี้แล้วใช้ตัวกรองดูใหม่ คราวนี้เน้นส่วนเปลี่ยนในแนวตั้ง จะเห็นว่าตัวกรองแนวตั้งเห็นค่าบวกลบชัดเจน
arr = np.array([[1,1,1,0,0,0],
[1,1,1,1,1,1],
[1,1,1,1,1,1],
[1,1,1,1,1,1],
[0,0,0,0,1,1],
[0,0,0,0,0,0]],
dtype=np.float32)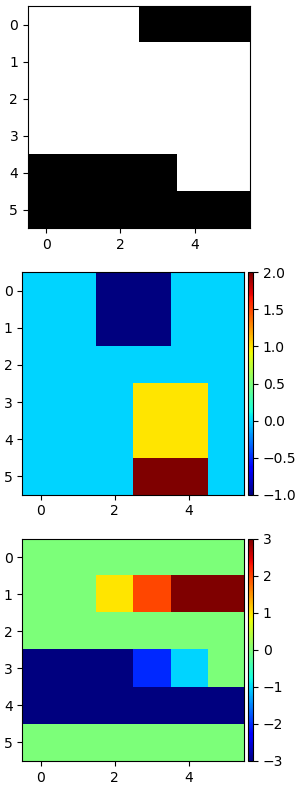
คราวนี้ลองเอามาใล้กับภาพที่เตรียมเอาไว้ดูบ้าง แม้จะเป็นภาพสีแต่เวลาค้นหาขอบมักจะแปลงเป็นขาวดำ ดังนั้นจะเปิดในโหมดขาวดำโดยใส่แฟล็ก cv2.IMREAD_GRAYSCALE ตอนอ่านภาพ

miku09c01.jpg
miku = cv2.imread('miku09c01.jpg',cv2.IMREAD_GRAYSCALE).astype(np.float32)
plt.figure(figsize=[6,11])
plt.subplot(311,xticks=[],yticks=[])
plt.imshow(miku,cmap='gray')
plt.colorbar(pad=0.01)
plt.subplot(312,xticks=[],yticks=[])
plt.imshow(cv2.filter2D(miku,-1,prewittx),cmap='copper')
plt.colorbar(pad=0.01)
plt.subplot(313,xticks=[],yticks=[])
plt.imshow(cv2.filter2D(miku,-1,prewitty),cmap='copper')
plt.colorbar(pad=0.01)
plt.tight_layout()
plt.show()
ผลที่ได้จะได้ภาพที่เน้นขอบในแนวตั้งและแนวนอนต่างกันไป
ตัวกรองโซเบล
ตัวกรองเพรวิตดังที่กล่าวมาข้างต้นเป็นรูปแบบตัวกรองอย่างง่ายๆซึ่งเป็นพื้นฐาน แต่ในทางปฏิบัติแล้วที่นิยมใช้จริงๆคือตัวกรองโซเบล (Sobel filter)
ข้อแตกต่างจากตัวกรองเพรวิตคือเพิ่มค่าตรงกลางจาก 1,-1 เป็น 2,-2 เพื่อเน้นตรงกลางมากขึ้น
ใน opencv ได้เตรียมฟังก์ชันสำหรับทำตัวกรองโซเบลไว้โดยไม่จำเป็นต้องสร้างเอง นั่นคือฟังก์ชัน cv2.Sobel()
| ลำดับ | ชื่อ | สิ่งที่ต้องใส่ | ชนิดข้อมูล |
|---|---|---|---|
| 1 | src | อาเรย์รูปภาพ | np.array |
| 2 | ddepth | ชนิดตัวแปรในอาเรย์ | flag (int) |
| 3 | dx | ขนาดตัวกรองแกน x | int |
| 4 | dy | ขนาดตัวกรองแกน y | int |
หากต้องการตัวกรองโซเบลแนวนอน (กรองขอบแนวตั้ง) ให้ใส่เป็น cv2.Sobel(ภาพ,-1,1,0) ถ้าต้องการตัวกรองในแนวตั้ง (กรองขอบแนวนอน) ให้ใส่ cv2.Sobel(ภาพ,-1,0,1)
ลองใช้กับภาพเดิมที่ใช้ในตัวอย่างที่แล้วแต่เปลี่ยนเป็นตัวกรองโซเบลแทน
miku = cv2.imread('miku09c01.jpg',0).astype(np.float32)
plt.figure(figsize=[6,11])
plt.subplot(311,xticks=[],yticks=[])
plt.imshow(miku,cmap='gray')
plt.colorbar(pad=0.01)
plt.subplot(312,xticks=[],yticks=[])
plt.imshow(cv2.Sobel(miku,-1,1,0),cmap='copper')
plt.colorbar(pad=0.01)
plt.subplot(313,xticks=[],yticks=[])
plt.imshow(cv2.Sobel(miku,-1,0,1),cmap='copper')
plt.colorbar(pad=0.01)
plt.tight_layout()
plt.show()
ตัวกรองชาร์
ใน opencv ยังมีเตรียมตัวกรองอีกอันที่คล้ายกับตัวกรองโซเบล คือตัวกรองชาร์ (Scharr filter) โดยฟังก์ชันcv2.Scharr()
วิธีการใช้คล้ายกับตัวกรองโซเบล ผลที่ได้ก็ใกล้เคียงกัน
ตัวอย่าง ลองใช้กับภาพเดิม
miku = cv2.imread('miku09c01.jpg',0).astype(np.float32)
plt.figure(figsize=[6,11])
plt.subplot(311,xticks=[],yticks=[])
plt.imshow(miku,cmap='gray')
plt.colorbar(pad=0.01)
plt.subplot(312,xticks=[],yticks=[])
plt.imshow(cv2.Scharr(miku,-1,1,0),cmap='copper')
plt.colorbar(pad=0.01)
plt.subplot(313,xticks=[],yticks=[])
plt.imshow(cv2.Scharr(miku,-1,0,1),cmap='copper')
plt.colorbar(pad=0.01)
plt.tight_layout()
plt.show()
ตัวกรองลาปลาเชียน
ทั้งตัวกรองเพรวิต, ตัวกรองโซเบล และตัวกรองชาร์ ซึ่งแนะนำมาในตัวอย่างที่ผ่านมานั้นจะเน้นขอบที่ปรากฏในแนวตั้งหรือแนวนอนอย่างใดอย่างหนึ่ง ส่วนกรณีที่ต้องการเน้นขอบทั้ง ๒ แนวเท่าๆกันนั้นอาจใช้ตัวกรองลาปลาเซียน (Laplacian filter)
จะเห็นว่ามีสมมาตรในแนวตั้งและแนวนอน
ตัวกรองลาปลาเซียนนี้มีที่มาจากตัวดำเนินการลาปลาเซียน ∇2 ซึ่งทำการคำนวณอนุพันธ์ย่อยอันดับที่สองของทั้งในแนวแกน x และ y
ต่อไปเป็นหลักการคำนวณซึ่งเป็นที่มาของตัวกรองลาปลาเซียน (ตรงนี้เป็นคณิตศาสตร์ระดับมหาวิทยาลัย ถ้าไม่สนใจรู้ลึกถึงขนาดนั้นอาจอ่านข้ามไปดูโค้ดได้เลย)
ให้ M(x,y) เป็นค่าความเข้มในตำแหน่ง (x,y) อนุพันธ์ย่อยทั้งในแกน x และ y อาจคำนวณได้โดย
จากนั้นคำนวณอนุพันธ์อันดับสอง
ลาปลาเซียนได้จากการเอาอนุพันธ์ย่อยอันดับสองทั้งหมดมารวมกัน ได้เป็น
ดังนั้นจึงได้ตัวกรองออกมาในลักษณะที่มี -4 ตรงกลาง และ +1 ที่บนล่างซ้ายขวา
ใน opencv มีฟังก์ชันสำหรับทำลาปลาเซียน คือ cv2.Laplacian()
ลองสร้างอาเรย์ง่ายๆขึ้นมาแล้วใช้ตัวกรองลาปลาเซียนกรองดู
arr = np.array([[0,1,1,1,0,0,0],
[0,1,1,1,1,0,0],
[0,0,1,1,1,1,0],
[0,0,1,1,1,1,0],
[0,0,1,1,1,1,0],
[0,0,0,0,1,0,0],
[0,0,0,0,0,0,0]],
dtype=np.float32)
plt.figure(figsize=[4,7])
plt.subplot(211)
plt.imshow(arr,cmap='gray')
plt.subplot(212)
plt.imshow(cv2.Laplacian(arr,-1),cmap='jet')
plt.colorbar(pad=0.01,aspect=40)
plt.tight_layout()
plt.show()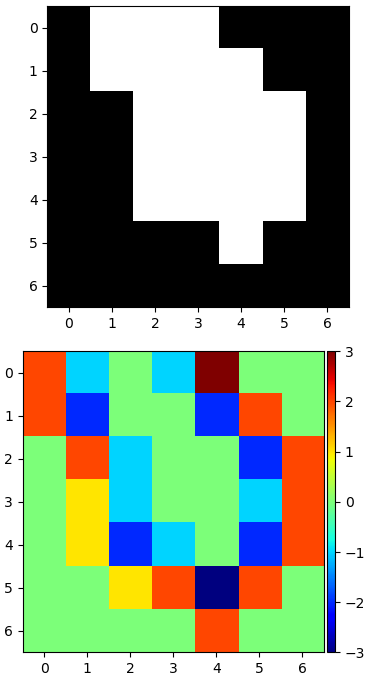
ผลลัพธ์จะเห็นว่าตรงที่เป็นจุดเปลี่ยนจะเป็นบวกและลบอย่างชัดเจนขึ้นมา ส่วนจุดที่ห่างไกลจากจุดเปลี่ยนจะเป็น 0 ดังนั้นส่วนขอบจึงถูกเน้นได้ด้วยตัวกรองนี้
คราวนี้ลองนำมาใช้กับภาพเดิมจากในตัวอย่างที่แล้ว
miku = cv2.imread('miku09c01.jpg',0).astype(np.float32)
plt.figure(figsize=[6,7.5])
plt.subplot(211,xticks=[],yticks=[])
plt.imshow(miku,cmap='gray')
plt.colorbar(pad=0.01)
plt.subplot(212,xticks=[],yticks=[])
plt.imshow(cv2.Laplacian(miku,-1),cmap='copper')
plt.colorbar(pad=0.01)
plt.tight_layout()
plt.show()
ผลที่ได้จะเน้นส่วนที่เป็นขอบไม่ว่าจะแนวไหน
ตัวกรองสร้างลายนูน
ตัวกรองอีกรูปแบบสำหรับทำเน้นขอบก็คือการทำเป็นลายนูน (emboss)
ตัวกรองสร้างลายนูนมีลักษณะแบบนี้
ลักษณะจะใกล้เคียงกับตัวกรองโซเบล แต่อยู่ในแนวเฉียง
ตัวอย่าง ใช้กับภาพเดิมจากตัวอย่างที่แล้ว
ใน opencv ไม่ได้เตรียมฟังก์ชันสำหรับสร้างลายนูนไว้โดยเฉพาะ จึงต้องสร้างตัวกรองขึ้นเองแล้วนำมาใช้ใน cv2.filter2D()
miku = cv2.imread('miku09c01.jpg',0).astype(np.float32)
emboss = np.array([[-2,-1,0],
[-1,1,1],
[0,1,2]])
plt.figure(figsize=[6,7.5])
plt.subplot(211,xticks=[],yticks=[])
plt.imshow(miku,cmap='gray')
plt.colorbar(pad=0.01)
plt.subplot(212,xticks=[],yticks=[])
plt.imshow(cv2.filter2D(miku,-1,emboss),cmap='copper')
plt.colorbar(pad=0.01)
plt.tight_layout()
plt.show()
เมื่อใช้ตัวกรองสร้างลายนูนแล้วก็ทำให้ภาพดูมีมิติขึ้นมา
วิธีการค้นหาขอบของคันนี
อีกวิธีที่นิยมใช้ในการค้นหาของก็คือการใช้วิธีการของคันนี (Canny) วิธีนี้ประกอบไปด้วยหลายขั้นตอนเพื่อทำการคัดกรองเอาส่วนที่เป็นขอบ ในนั้นมีการใช้ตัวกรองเกาส์และตัวกรองโซเบลเข้าร่วมด้วย
ฟังก์ชันสำหรับใช้ตัวกรองคันนีใน opencv คือ cv2.Canny() ค่าที่ต้องใส่ในฟังก์ชันเป็นดังนี้
| ลำดับ | ชื่อ | สิ่งที่ต้องใส่ | ชนิดข้อมูล |
|---|---|---|---|
| 1 | image | อาเรย์รูปภาพ | np.array |
| 2 | threshold1 | ค่าจุดเปลี่ยน 1 | int |
| 2 | threshold2 | ค่าจุดเปลี่ยน 2 | int |
ในที่นี้ threshold1 และ threshold2 คือค่าจุดเปลี่ยนสำหรับคัดกรองว่าตรงไหนควรจะเป็นขอบ โดยภายในกระบวนการของวิธีนี้จะมีการคำนวณค่าความน่าจะเป็นขอบของแต่ละจุดอยู่ จากนั้นค่านั้นจะถูกนำมาพิจารณาโดยดูค่ากรองต่ำสุดและสูงสุดดังนี้
| จุดที่ค่าต่ำกว่าค่ากรองต่ำสุด | 0 (ไม่ใช่ขอบ) | |
| จุดที่ค่าอยู่ระหว่าง ค่ากรองต่ำสุดและสูงสุด |
ถ้าไม่มีจุดที่ค่าสูงกว่าค่ากรองสูงสุดอยู่ข้างๆ | |
| ถ้ามีจุดที่ค่าสูงกว่าค่ากรองสูงสุดอยู่ข้างๆ | 255 (เป็นขอบ) | |
| จุดที่ค่าสูงกว่าค่ากรองสูงสุด | ||
threshold1 และ threshold2 ที่ใส่ลงไปนี้ตัวหนึ่งจะเป็นค่ากรองสูงสุด อีกตัวเป็นค่ากรองต่ำสุด จะใส่ตัวไหนเป็นค่ามากกว่าก็ได้ ใส่สลับกันก็ไม่มีผลต่างกัน
อาเรย์ภาพที่ใช้กับตัวกรองนี้ใช้เป็น uint8 เท่านั้น ไม่ต้องแปลงเป็น float32 เหมือนอย่างวิธีอื่นที่กล่าวมาข้างต้น
ผลที่ได้จากตัวกรองคันนีจะออกมาเป็นค่าที่มีแค่ ๒ ค่า คือส่วนที่เป็นขอบและส่วนที่ไม่เป็นขอบเท่านั้น โดยส่วนที่เป็นขอบจะได้ค่าเป็น 255 และที่ไม่ใช่ขอบจะเป็น 0 ชนิดข้อมูลที่ได้ก็จะเป็น uint8 เสมอ
ลองใช้กับภาพเดิมจากในตัวอย่างที่แล้ว
miku = cv2.imread('miku09c01.jpg',0)
plt.figure(figsize=[6,9])
plt.subplot(211,xticks=[],yticks=[])
plt.imshow(miku,cmap='gray')
plt.subplot(212,xticks=[],yticks=[])
plt.imshow(cv2.Canny(miku,50,100),cmap='copper')
plt.tight_layout()
plt.show()
ลองอีกตัวอย่างเพื่อเปรียบเทียบผลลัพธ์ระหว่างค่าจุดเปลี่ยนต่างกัน

rin09c01.jpg
rin = cv2.imread('rin09c01.jpg',0)
plt.figure(figsize=[6,7.5])
thr = [(0,50),(0,100),(50,100),(50,150),(100,150),(100,200)]
for i in range(6):
plt.subplot(321+i,xticks=[],yticks=[],title='threshold = (%d,%d)'%thr[i])
carin = cv2.Canny(rin,thr[i][0],thr[i][1])
plt.imshow(carin,cmap='copper')
plt.tight_layout()
plt.show()
จะเห็นว่ายิ่งกำหนดค่าต่ำก็จะยิ่งคัดกรองเอาจุดที่เป็นขอบมามาก แต่ก็มีส่วนที่อาจไม่ได้ต้องการปนมามากขึ้น
อ่านบทถัดไป >> บทที่ ๑๐
-----------------------------------------
囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧囧
หมวดหมู่
-- คอมพิวเตอร์ >> เขียนโปรแกรม >> opencv-- คอมพิวเตอร์ >> เขียนโปรแกรม >> python >> numpy
-- คอมพิวเตอร์ >> เขียนโปรแกรม >> python >> matplotlib